- Membres
- Nédia KAMECH
Nédia KAMECH
Mes travaux de recherche portent sur les structures secondaire et tridimensionnelle des peptides et des neuropeptides.
présentation chronologique
- Les peptides antimicrobiens de batraciens comme les grenouilles du genre Rana sont des molécules qui interviennent dans la défense des organismes et des plantes et qui ont pour cible principale la membrane cellulaire des microorganismes, à savoir bactéries, virus ou champignons microscopiques et interfèrent ainsi avec les voies métaboliques intracellulaires.
Une autre catégorie de peptides comme les neuropeptides est impliquée dans le développement, les fonctions métaboliques ou la reproduction et module les circuits neuronaux qui interviennent dans les comportements adaptatifs de l'animal. Chez l'écrevisse, la CHH ou crustacean hyperglycemic hormone, a la particularité d'être synthétisée sous deux formes isomères, ne différant que par la chiralité d'un acide aminé de conformation L ou D. Ces isomères ont des activités différentes et agissent sur des cibles tissulaires différentes.
Chez l'anguille, de nombreux neuropeptides interviennent dans l'axe gonadotrope comme la gonadolibérine, la dopamine ou les kisspeptines. Ces peptides hormonaux exercent leurs fonctions en interagissant avec des récepteurs membranaires appartenant le plus souvent aux récepteurs couplés aux protéines G qui ont une structure tridimensionnelle très conservée. Le GnRH associated peptide, GAP, situé dans la région carboxy-terminale de la preproGnRH (Gonadotropin-releasing hormone) possède un motif de repliement tridimensionnel en Hélice-Boucle-Hélice (Helix Loop Helix, HLH) très conservé dans le règne des vertébrés. Ceci suggère un rôle important du GAP dans les fonctions neuroendocrines ( http://www.anr-nemo.org/fr). D'autres neuropeptides comme les tachykinines agissent sur la reproduction via les recepteurs à GnRH.
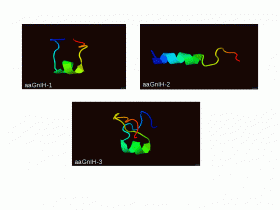
Récemment l'identification dans notre laboratoire de 3 peptides paralogues de la la GnIH chez l'anguille européenne a notamment permis de prédire une structure essentiellement en hélice alpha bordée de 2 régions en boucles côtés N et C-terminaux avec une conformation originale tubulaire pour la GnIH-2.
J' enseigne la Biochimie, la Biologie moléculaire et la Bioinformatique à Sorbonne Université SU sur le campus Pierre et Marie Curie à Paris. Cet enseignement est destiné à des étudiants de licence et de master, ainsi qu'à des chercheurs du public et du privé dans le cadre de la formation continue.
Mon enseignement en Biochimie-Biologie moléculaire porte sur la structure des protéines et sur leur biosynthèse au sein de la cellule. Il porte aussi sur les techniques de clonage et de purification qui permettent d'obtenir des protéines recombinantes.
En Bioinformatique, j'enseigne les langages de programmation Java, C, C++ et Python ; j'ai par ailleurs participé à un enseignement en Analyse d'Images pendant plusieurs années. Enfin, les nombreux outils logiciels utilisés en Biochimie pour l'analyse des protéines ont fait l'objet d'un enseignement dont j'ai été responsable dans le cadre de la formation continue de l'université pendant plusieurs années.
Je m'implique également dans l'intégration des étudiants à l'université en étant la référente de certains d'entre eux et en participant à un enseignement en licence (1ère et 3 ème années) qui les incite à réfléchir à leur cursus et à leur orientation au sein de SU. J'enseigne aussi en " Insertion Professionnelle" aux étudiants du master Biologie Moléculaire et Cellulaire de Sorbonne Université.
J'ai également participé 3 années consécutives à la mise en place d'un enseignement pluridisciplinaire d' Histoire et de Philosophie des sciences au master BMC en me focalisant plus particulièrement sur les articulations Biologie / Société et Biologie / Histoire.
Enfin, dans le cadre d'un enseignement de kinesthésie, intitulé l'Apprentissage en mouvement, soutenu par un IDEX Sorbonne Université (2013-2016) et avec des collègues physiciens, littéraires et du centre sportif du campus clignancourt de Sorbonne Université, nous avons monté un atelier Danse et Biologie à destination des étudiants de licence; un blog décrit cette expérience pluridisciplinaire http://kinesthesie.canalblog.com .
Je poursuis actuellement ce travail associant Biologie et Danse puisqu'un enseignement intitulé Biogeste est proposé depuis 2015 aux étudiants de premère année de licence de Biologie dans le cadre des ateliers de recherche encadrée. Cette expérience pédagogique a fait l'objet d'une publication (Questions de Pédagogie dans l’Enseignement Supérieur 2015 , http://www.colloque-pedagogie.org/node/751 pages 730-739).
Publication en Pédagogie dans l'Enseignement Supérieur
Emmanuel Rollinde, Isabella Montersino, Philippe Brunet, Nédia Kamech, Françoise Loakes-Gouju, Stefano Cossara, Barbara Le Lan.
Apprentissage en mouvement, un projet pluridisciplinaire de Sorbonne Universités autour de la kinesthésie. Actes du VIII e colloque QPES, Questions de Pédagogie dans l’Enseignement Supérieur, Brest 17 -19 juin 2015, 730-739. http://www.colloque-pedagogie.org/node/751
pubmed
Mes publications
2020
-
Maugars, G. , J. Pasquier, C. Atkinson, A.-G. Lafont, A. Campo, Nedia Kamech, B. Lefranc, J. Leprince, Sylvie Dufour, et K. Rousseau. 2020. « Gonadotropin-Inhibitory Hormone In Teleosts: New Insights From A Basal Representative, The Eel ». General And Comparative Endocrinology 287: 113350. doi:10.1016/j.ygcen.2019.113350. https://linkinghub.elsevier.com/retrieve/pii/S0016648019303296.
2018
-
Campo, Aurora , Anne-Gaelle Lafont, Benjamin Lefranc, Jérôme Leprince, Hervé Tostivint, Nedia Kamech, Sylvie Dufour, et Karine Rousseau. 2018. « Tachykinin-3 Genes And Peptides Characterized In A Basal Teleost, The European Eel: Evolutionary Perspective And Pituitary Role. ». Frontiers In Endocrinology 9: 304.
 Campo et al 2018.pdf (3.75 Mo)
Campo et al 2018.pdf (3.75 Mo)
2017
-
Sirkin, Daniela IPérez, Anne-Gaelle Lafont, Nedia Kamech, Gustavo M Somoza, Paula G Vissio, et Sylvie Dufour. 2017. « Conservation Of Three-Dimensional Helix-Loop-Helix Structure Through The Vertebrate Lineage Reopens The Cold Case Of Gonadotropin-Releasing Hormone-Associated Peptide ». Frontiers In Endocrinology 8: 207. doi:10.3389/fendo.2017.00207. https://www.frontiersin.org/article/10.3389/fendo.2017.00207.
2014
-
Pasquier, J , Nedia Kamech, Anne-Gaelle Lafont, H Vaudry, Karine Rousseau, et Sylvie Dufour. 2014. « Molecular Evolution Of Gpcrs: Kisspeptin/Kisspeptin Receptors. ». J. Mol. Endocrinol. 52 (3): 101-117.
 PasquierJME2014.pdf (345.61 Ko)
PasquierJME2014.pdf (345.61 Ko) -
Simunic, J , D Petrov, T Bouceba, Nedia Kamech, M Benincasa, et D Juretic. 2014. « Trichoplaxin - A New Membrane-Active Antimicrobial Peptide From Placozoan Cdna. ». Biochim. Biophys. Acta 1838 (5): 1430-1438.