Lorane Le Franc soutiendra sa thèse de doctorat intitulée :
Devant le jury :
Régulation épigénétique du développement : approche eco-evo-devo des m6A épitranscriptomes chez l’huître Crassostrea gigas
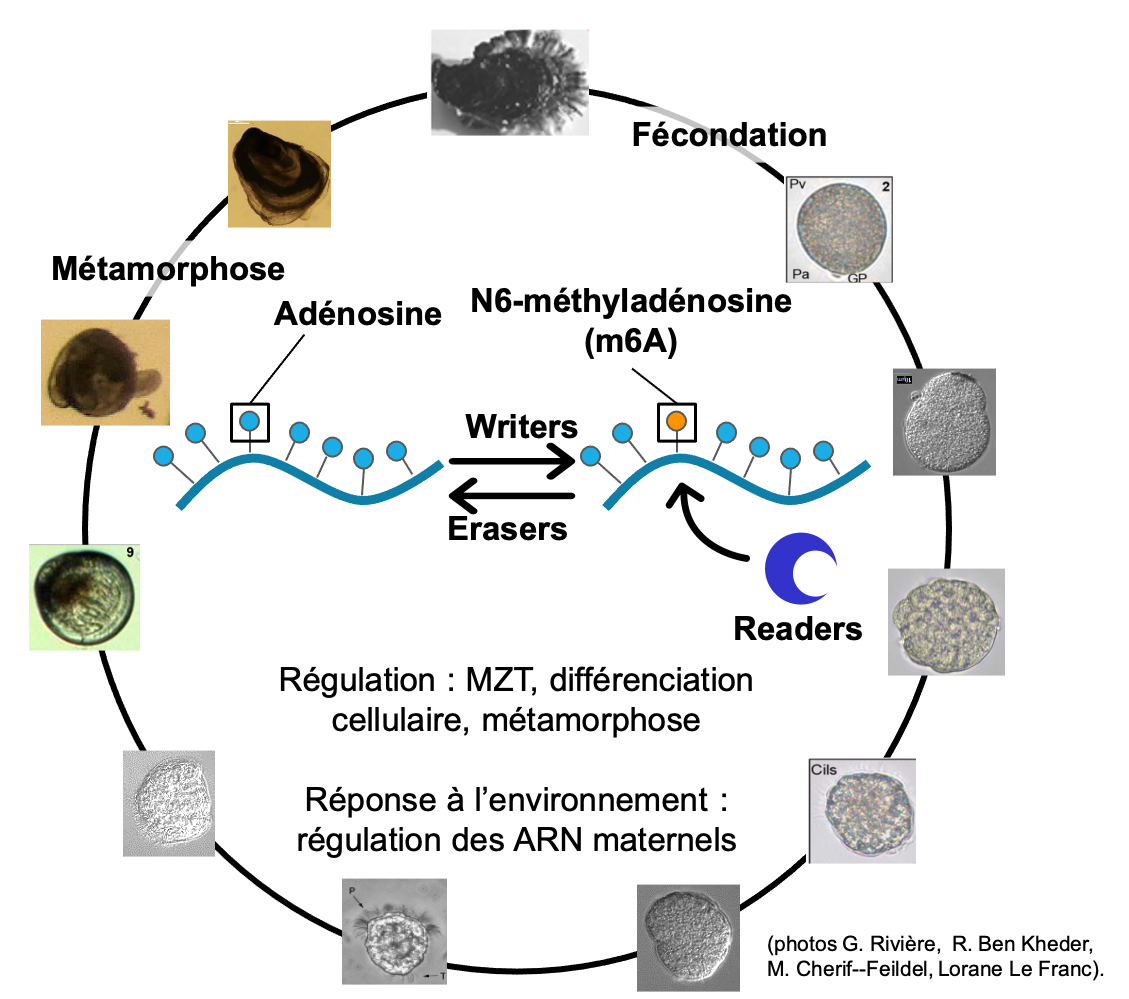
La méthylation de l’ARN N6 -méthyladénosine (m6A) est un nouveau mécanisme de régulation de l’expression des gènes via son implication dans l’épissage, la traduction et la stabilité des transcrits. Cette voie épitranscriptomique, composée de writers, d’erasers, et des readers, régule des étapes clés du développement comme la MZT et la différenciation cellulaire. Cependant, cette voie épitranscriptomique, influencée par l’environnement, montre des différences entre les Vertébrés et Ecdysozoaires étudiés et pose la question de sa conservation dans l’ensemble des Bilatériens et particulièrement chez les Lophotrochozoaires. L’étude de la conservation de la voie épitranscriptomique chez C. gigas a été réalisée par détection de la m6A par dotblot et spectrométrie de masse. Des orthologues de l’ensemble des acteurs de la machinerie ont été caractérisés in silico, et la présence de readers fonctionnels confirmée par RNA pull down/LC-MS2 . L’analyse des méthylomes développementaux obtenus par MeRIP-seq montre une dynamique du niveau et de la localisation de la m6A au cours du développement de l’huître. Ces résultats montrent une régulation de la segmentation, de la gastrulation, de la différenciation tissulaire et de la métamorphose par la m6A, chez l’huître creuse. Enfin l’analyse de méthylomes ovocytaires dans des conditions environnementales contrastées démontre une fonction de transmission de trait de vie portée par la m6A chez l’huître. L’ensemble de ce travail a permis de démontrer la conservation du rôle développemental des épitranscriptomes m6A et de leur rôle de vecteur intergénérationnel de traits de vie pour la première fois, à notre connaissance, chez un Lophotrochozoaire.
Mots clés : m6A, développement, huître, ARN, méthylation, épitranscriptomique
Abstract:
Epigenetic regulation of development: eco-evo-devo approach of m6A epitranscriptomes in the oyster Crassostrea gigas
The RNA modification N6 -methyladenosine (m6A) is a newly described regulatory layer of gene expression involved in RNA splicing, translation and stability. This epitranscriptomic pathway, composed of writers, erasers and readers, regulates key developmental steps such as MZT and cell differentiation. However, this epitranscriptomic mechanism, which is under environmental influence, shows differences between the Vertebrates and Ecdysozoans examined, which raise the question of the general conservation of an epitranscriptomic pathway in Bilaterian and especially in Lophotrochozoans. Therefore, we investigated the conservation of an m6A pathway in C. gigas by detection of m6A RNA using dotblot and mass spectrometry. Orthologues of all types of actors of the m6A machinery were characterized in silico, and the presence of functional readers was assessed by RNA pull-down/LC-MS2. The profiling of embryo-larval m6A methylomes obtained by MeRIPseq showed that the m6A level and localization were dynamic throughout oyster development. These results show a regulation of oyster segmentation, gastrulation, tissue differentiation and metamorphosis by the m6A. Finally, the study of oocyte methylomes in contrasted environmental conditions demonstrates a life trait transmission function carried by m6A in the oyster. This work demonstrates the conservation of the developmental function of m6A epitranscriptomes and their role as a vector of intergenerational life traits for the first time, to our knowledge, in a Lophotrochozoan. Keywords: m6A, development, oyster, RNA, methylation, epitranscriptomic
« Régulation épigénétique du développement : approche eco-evo-devo des m6A épitranscriptomes chez l’huître Crassostrea gigas »
Date : Mardi 18 mai 2021, à 14H
Lieu : Université Caen Normandie (Campus 1, amphithéâtre Jacquet)Devant le jury :
- M. Marc Graille, Directreur de recherche, Ecole polytechnique, CNRS - Rapporteur
- Mme Céline Cosseau, Maître de conférences HDR, Université Perpignan Via Domitia - Rapporteur
- Mme Céline Bousquet-Antonelli, Directrice de recherche, Université Perpigan Via Domitia - Examinateur
- Mme Charlotte Corporeau, Chargée de recherche HDR, Ifremer Bretagne - Examinateur
- M. Guillaume Rivière, Maître de conférences HDR, Université Caen Normandie/BOREA - Directeur de thèse
- M. Pascal Favrel, Professeur, Université Caen Normandie/BOREA - Co-directeur de thèse
Régulation épigénétique du développement : approche eco-evo-devo des m6A épitranscriptomes chez l’huître Crassostrea gigas
La méthylation de l’ARN N6 -méthyladénosine (m6A) est un nouveau mécanisme de régulation de l’expression des gènes via son implication dans l’épissage, la traduction et la stabilité des transcrits. Cette voie épitranscriptomique, composée de writers, d’erasers, et des readers, régule des étapes clés du développement comme la MZT et la différenciation cellulaire. Cependant, cette voie épitranscriptomique, influencée par l’environnement, montre des différences entre les Vertébrés et Ecdysozoaires étudiés et pose la question de sa conservation dans l’ensemble des Bilatériens et particulièrement chez les Lophotrochozoaires. L’étude de la conservation de la voie épitranscriptomique chez C. gigas a été réalisée par détection de la m6A par dotblot et spectrométrie de masse. Des orthologues de l’ensemble des acteurs de la machinerie ont été caractérisés in silico, et la présence de readers fonctionnels confirmée par RNA pull down/LC-MS2 . L’analyse des méthylomes développementaux obtenus par MeRIP-seq montre une dynamique du niveau et de la localisation de la m6A au cours du développement de l’huître. Ces résultats montrent une régulation de la segmentation, de la gastrulation, de la différenciation tissulaire et de la métamorphose par la m6A, chez l’huître creuse. Enfin l’analyse de méthylomes ovocytaires dans des conditions environnementales contrastées démontre une fonction de transmission de trait de vie portée par la m6A chez l’huître. L’ensemble de ce travail a permis de démontrer la conservation du rôle développemental des épitranscriptomes m6A et de leur rôle de vecteur intergénérationnel de traits de vie pour la première fois, à notre connaissance, chez un Lophotrochozoaire.
Mots clés : m6A, développement, huître, ARN, méthylation, épitranscriptomique
Abstract:
Epigenetic regulation of development: eco-evo-devo approach of m6A epitranscriptomes in the oyster Crassostrea gigas
The RNA modification N6 -methyladenosine (m6A) is a newly described regulatory layer of gene expression involved in RNA splicing, translation and stability. This epitranscriptomic pathway, composed of writers, erasers and readers, regulates key developmental steps such as MZT and cell differentiation. However, this epitranscriptomic mechanism, which is under environmental influence, shows differences between the Vertebrates and Ecdysozoans examined, which raise the question of the general conservation of an epitranscriptomic pathway in Bilaterian and especially in Lophotrochozoans. Therefore, we investigated the conservation of an m6A pathway in C. gigas by detection of m6A RNA using dotblot and mass spectrometry. Orthologues of all types of actors of the m6A machinery were characterized in silico, and the presence of functional readers was assessed by RNA pull-down/LC-MS2. The profiling of embryo-larval m6A methylomes obtained by MeRIPseq showed that the m6A level and localization were dynamic throughout oyster development. These results show a regulation of oyster segmentation, gastrulation, tissue differentiation and metamorphosis by the m6A. Finally, the study of oocyte methylomes in contrasted environmental conditions demonstrates a life trait transmission function carried by m6A in the oyster. This work demonstrates the conservation of the developmental function of m6A epitranscriptomes and their role as a vector of intergenerational life traits for the first time, to our knowledge, in a Lophotrochozoan. Keywords: m6A, development, oyster, RNA, methylation, epitranscriptomic