Offre Post-Doctorat 12 mois | Structure de la cHromatine, Environnement et Méthylation des Acides nucléiques chez l’huître (SCHEMAh), Caen
Offre Post-Doctorat 12 mois | Structure de la cHromatine, Environnement et Méthylation des Acides nucléiques chez l’huître (SCHEMAh), Caen
Supervision / contact : Guillaume Rivière, PHD HDR, guillaume.riviere@unicaen.fr ,+33231565113
Structure d'accueil : Laboratoire UMR BOREA, MNHN, CNRS 8067, SU, IRD 207, UCN, UA ; site de Caen
Début du contrat : A définir, au plus tôt le 1e Février 2022
Durée du contrat : 12 mois
Date limite de candidature : 17 décembre 2021
Contexte général :
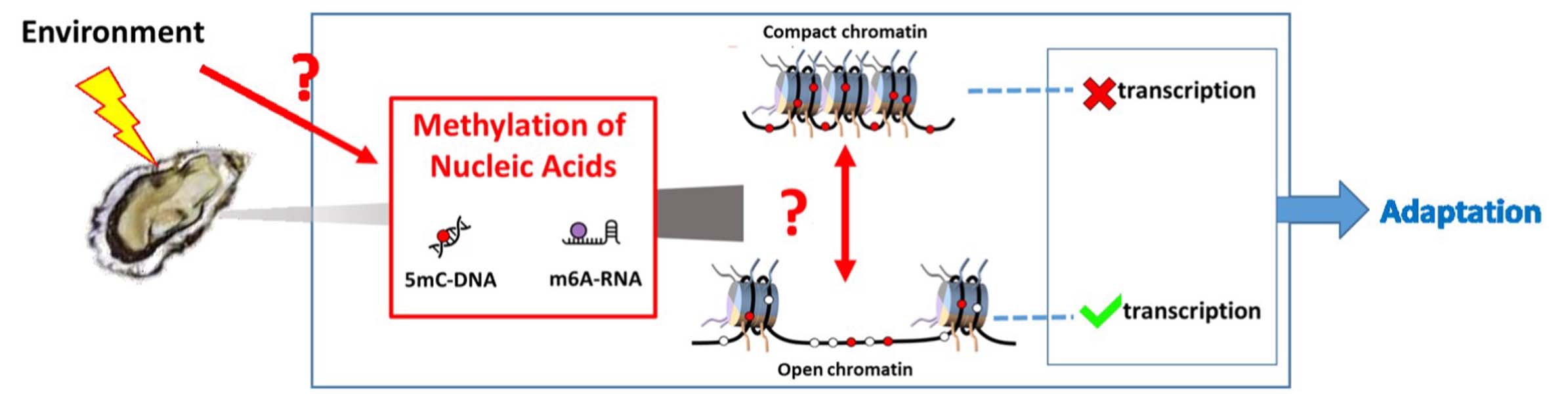
Les organismes s’adaptent aux changements de l’environnement notamment grâce à la modification de l’expression de leurs gènes permettant une réponse physiologique appropriée. Mais quel(s) mécanisme(s) régi(ssen)t-il(s) l’orientation de cette réponse ? Cette question est cruciale dans le contexte du changement global mais reste largement énigmatique. Néanmoins, des données récentes suggèrent que les paramètres environnementaux déterminent l’expression des gènes en façonnant l’architecture du matériel génétique c’est à dire la structure de la chromatine, via la méthylation des acides nucléiques (ADN et ARN). En effet, ce mécanisme épigénétique prévalent favorise la condensation de la chromatine (méthylation 5mC-ADN) et permet le verrouillage des chromosomes surnuméraires (méthylation m6A-ARN) chez les Mammifères. L’huître creuse Crassostrea gigas est la ressource conchylicole la plus importante de Normandie, et son écologie la rend particulièrement exposée aux changements de l’environnement (marées, saisons, changement global). Cependant, bien que C. gigas présente de la 5mC-ADN et de la m6A-ARN, et que les huîtres triploïdes soient robustes voire fertiles, l’influence de la méthylation des acides nucléiques sur la structure de la chromatine chez l’huître reste inconnue à ce jour. Le projet SCHEMAh propose pour la première fois de comprendre si et comment l’environnement contrôle la structure du matériel génétique via la méthylation des acides nucléiques chez le modèle ‘huître’, et ainsi l’expression des gènes permettant son adaptation. Par des approches innovantes combinées aux niveaux moléculaires (Chromatin Accessibility Assay, ATAC-seq, séquençage Nanopore) et morphologiques (histologie quantitative et microscopie électronique 3D), le projet SCHEMAh explorera d’une part le lien entre méthylation de l’ADN (5mC) et structure de la chromatine, et d’autre part l’existence d’ARNs non codants m6A-methylés (m6A-carRNAs) qui pourraient participer à la compensation du dosage chromosomique aberrant des huîtres triploides. Les connaissances fondamentales pionnières apportées par ce projet constituent un enjeu important pour comprendre l’adaptation de l’huître C. gigas, un modèle d'intérêt scientifique majeur et maillon clé de l'écosystème littoral normand, et plus généralement des espèces, face au changement global.
Missions :
La personne recrutée aura pour mission principale de réaliser l’analyse bioinformatique des données issues des méthodologies de séquençage haut-débit mises en œuvre dans le cadre du projet (RNA-seq, Methyl-seq, m6A-RNA-seq, ATAC-seq …). La personne recrutée, d’une manière plus générale, participera à l’ensemble du projet SCHEMAh et plus spécifiquement à la génération du matériel biologique (traitements d’animaux au laboratoire, dissections, extractions d’acides nucléiques) et à la construction des librairies de séquençage ADN et ARN, dont une partie pourra être réalisée en externe. La personne recrutée pourra également participer au développement de la cartographie directe à haute résolution de modifications d’acides nucléiques par séquençage Nanopore.
Compétences attendues :
Le/la candidat(e) sera titulaire d’un doctorat en sciences de la vie orienté épigénomique, génomique, transcriptomique ou biologie moléculaire et bioinformatique. Il/elle et possèdera une bonne maîtrise théorique dans l’ensemble de ces domaines. Le/la candidat(e) devra avoir une solide expérience en analyse bioinformatique de données de séquençage NGS, ainsi que des compétences en biologie moléculaire à la paillasse. Une expérience ou une connaissance de modèles marins, idéalement dans un contexte écophysiologique ou de challenge environnemental, serait un plus mais n’est pas strictement requise. Intégré au sein d’une équipe pluridisciplinaire, le/la candidat(e) devra être curieux et capable de travailler au sein d’un groupe composé d’enseignants-chercheurs, de chercheurs, d’ingénieurs, de doctorants et d’étudiants en Master. A ce titre, un niveau de français ou d’anglais permettant une communication fluide est indispensable.
guillaume.riviere@unicaen.fr
objet : [SCHEMAh] postdoc